Phosphoglyceratmutase - Phosphoglycerate mutase
| Phosphoglycerat-Mutase-Familie | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Kennungen | |||||||||||
| Symbol | PGAM | ||||||||||
| Pfam | PF00300 | ||||||||||
| InterPro | IPR013078 | ||||||||||
| PROSITE | PDOC00158 | ||||||||||
| SCOP2 | 3 pgm / SCOPe / SUPFAM | ||||||||||
| |||||||||||
| Phosphoglyceratmutase 1 (Gehirn) | |||||||
|---|---|---|---|---|---|---|---|
 | |||||||
| Kennungen | |||||||
| Symbol | PGAM1 | ||||||
| Alt. Symbole | PGAMA | ||||||
| NCBI-Gen | 5223 | ||||||
| HGNC | 8888 | ||||||
| OMIM | 172250 | ||||||
| RefSeq | NM_002629 | ||||||
| UniProt | P18669 | ||||||
| Andere Daten | |||||||
| EG-Nummer | 5.4.2.11 | ||||||
| Ort | Chr. 10 q25.3 | ||||||
| |||||||
| Phosphoglyceratmutase 2 (Muskel) | |||||||
|---|---|---|---|---|---|---|---|
| Kennungen | |||||||
| Symbol | PGAM2 | ||||||
| NCBI-Gen | 5224 | ||||||
| HGNC | 8889 | ||||||
| OMIM | 261670 | ||||||
| RefSeq | NM_000290 | ||||||
| UniProt | P15259 | ||||||
| Andere Daten | |||||||
| EG-Nummer | 5.4.2.11 | ||||||
| Ort | Chr. 7 p13-p12 | ||||||
| |||||||
- Dieses Enzym ist nicht mit Bisphosphoglycerat-Mutase zu verwechseln , die die Umwandlung von 1,3-Bisphosphoglycerat zu 2,3-Bisphosphoglycerat katalysiert .
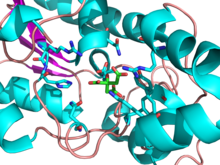
Phosphoglyceratmutase (PGM) ist ein beliebiges Enzym , das Schritt 8 der Glykolyse katalysiert . Sie katalysieren den internen Transfer einer Phosphatgruppe von C-3 zu C-2, was zur Umwandlung von 3-Phosphoglycerat (3PG) zu 2-Phosphoglycerat (2PG) über ein 2,3-Bisphosphoglycerat-Zwischenprodukt führt. Diese Enzyme sind kategorisiert in die zwei verschiedene Klassen von beiden Cofaktor -abhängigen (dPGM) oder Cofaktor-unabhängige (IPGM). Das dPGM-Enzym ( EC 5.4.2.11 ) besteht aus ungefähr 250 Aminosäuren und kommt in allen Wirbeltieren sowie in einigen Wirbellosen, Pilzen und Bakterien vor. Die iPGM- Klasse ( EC 5.4.2.12 ) kommt in allen Pflanzen und Algen sowie in einigen Wirbellosen, Pilzen und grampositiven Bakterien vor. Diese Klasse von PGM-Enzymen hat dieselbe Superfamilie wie alkalische Phosphatase .
Mechanismus
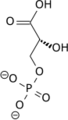
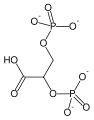
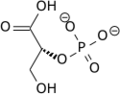
PGM ist ein Isomeraseenzym , das effektiv eine Phosphatgruppe (PO 4 3− ) vom C-3-Kohlenstoff von 3-Phosphoglycerat auf den C-2-Kohlenstoff überträgt, der 2-Phosphoglycerat bildet . Es gibt insgesamt drei Reaktionen, die dPGM katalysieren kann: eine Mutase-Reaktion, die zur Umwandlung von 3PG in 2PG und umgekehrt führt, eine Phosphatase-Reaktion, die Phosphoglycerat aus 2,3-Bisphosphoglycerat erzeugt, und eine Synthase-Reaktion, die 2,3-Bisphosphoglycerat aus 1 erzeugt 3-Bisphosphoglycerat ähnlich dem Enzym Bisphosphoglyceratmutase. Kinetische und strukturelle Studien haben nachgewiesen, dass dPGM und zeigen Bisphosphoglyceratmutase sind paraloge Strukturen. Beide Enzyme sind in der Superfamilie enthalten , die auch den Phosphataseanteil von Phosphofructokinase 2 und Prostatasäurephosphatase enthält .
Die katalysierte Mutase-Reaktion beinhaltet zwei getrennte Phosphorylgruppen und das Endphosphat auf dem 2-Kohlenstoff ist nicht das gleiche Phosphat, das aus dem 3-Kohlenstoff entfernt wurde.
Im Ausgangszustand des cofaktorabhängigen Enzyms enthält das aktive Zentrum einen Phosphohistidinkomplex, der durch Phosphorylierung eines spezifischen Histidinrests gebildet wird. Wenn 3-Phosphoglycerat in das aktive Zentrum eintritt , wird der Phosphohistidinkomplex so positioniert, dass der Transfer von Phosphat vom Enzym zum Substrat C-2 erleichtert wird, wodurch ein 2,3-Bisphosphoglycerat- Zwischenprodukt entsteht.
Dephosphorylierung des Enzym Histidin betätigt eine lokale allosterische Änderung der Enzym Konfiguration , die jetzt richtet das Substrate 3 C-Phosphatgruppe mit Enzyme aktiven Stelle Histidin und Phosphat - Transfer erleichtert die Rückkehr Enzym in seinen anfänglichen phosphorylierten Zustand und Freigeben Produkt 2-Phosphoglycerat . 2,3-Bisphosphoglycerat wird als Cofaktor für dPGM benötigt. Im Gegensatz dazu ist die iPGM-Klasse unabhängig von 2,3-Bisphosphoglycerat und katalysiert den intramolekularen Transfer der Phosphatgruppe auf Monophosphoglycerate unter Verwendung eines Phosphoserin-Zwischenprodukts.
Reaktionszusammenfassung
3PG + P-Enzym → 2,3BPG + Enzym → 2PG + P-Enzym
3-phosphoglycerate intermediate 2-phosphoglycerate
ΔG ° '= + 1,1 kcal / mol
Isozyme
Phosphoglyceratmutase existiert hauptsächlich als Dimer von zwei entweder identischen oder eng verwandten Untereinheiten von etwa 32 kDa. Das Enzym kommt in Organismen vor, die durch Homo sapiens so einfach wie Hefe sind, und seine Struktur ist durchgehend hoch konserviert. (Hefe-PGM ~ 74% konserviert gegenüber Säugetierform).
Bei Säugetieren scheinen die Enzymuntereinheiten entweder eine von Muskeln abgeleitete Form (m-Typ) oder ein anderes Gewebe (b-Typ für Gehirn, bei dem das b-Isozym ursprünglich isoliert wurde) zu sein. Das als Dimer vorhandene Enzym weist dann 3 Isozyme auf, je nachdem, welche Untereinheit das gesamte Molekül bildet (mm, bb oder mb). Der mm-Typ kommt fast ausschließlich in glatten Muskeln vor. Das mb-Isozym befindet sich im Herz- und Skelettmuskel und der bb-Typ im übrigen Gewebe. Während alle drei Isozyme in jedem Gewebe gefunden werden können, basieren die obigen Verteilungen auf der Prävalenz in jedem.
Interaktive Wegkarte
Klicken Sie unten auf Gene, Proteine und Metaboliten, um auf die entsprechenden Artikel zu verlinken.
Verordnung
Phosphoglyceratmutase hat eine kleine positive freie Gibbs-Energie und diese Reaktion verläuft leicht in beide Richtungen. Da es sich um eine reversible Reaktion handelt, sind hier keine wichtigen Regulationsmechanismen oder Regulationsschemata für den glykolytischen Weg vorhanden.
Anionische Moleküle wie Vanadat , Acetat , Chloridion , Phosphat , 2-Phosphoglykolat und N- [Tris (hydroxymethyl) methyl-2-amino] ethansulfonat sind bekannte Inhibitoren der Mutaseaktivität von dPGM. Studien haben gezeigt, dass dPGM empfindlich gegenüber Änderungen der Ionenkonzentration ist, wobei steigende Salzkonzentrationen zur Aktivierung der Phosphataseaktivität des Enzyms führen, während seine Mutaseaktivität gehemmt wird. Bestimmte Salze wie KCl sind als kompetitive Inhibitoren in Bezug auf die 2-Phosphoglycerat- und Mutaseaktivität bekannt. Sowohl Phosphat als auch 2-Phosphoglykolat sind kompetitive Inhibitoren der Mutaseaktivität in Bezug auf die Substrate 2-Phosphoglycerat und 2,3-Bisphosphoglycerat.
Klinische Bedeutung
Beim Menschen befindet sich das PGAM2-Gen, das dieses Enzym codiert, auf dem kurzen Arm von Chromosom 7.
Ein Mangel an Phosphoglyceratmutase verursacht eine Glykogenspeicherkrankheit Typ X , eine seltene autosomal rezessive genetische Störung mit Symptomen von leicht bis mittelschwer; wird nicht als lebensbedrohlich angesehen und kann mit Änderungen im Lebensstil bewältigt werden. Dies stellt eine metabolische Myopathie dar und ist eine der vielen Formen von Syndromen, die früher als Muskeldystrophie bezeichnet wurden. Ein PGAM1-Mangel betrifft die Leber, während ein PGAM2-Mangel den Muskel betrifft.
Der Beginn wird im Allgemeinen als Kindheit bis zum frühen Erwachsenen bezeichnet, obwohl einige, die möglicherweise leicht von der Störung betroffen sind, möglicherweise nicht wissen, dass sie diese haben. Patienten mit PGAM-Mangel sind normalerweise asymptomatisch, es sei denn, sie unternehmen kurze, anstrengende Anstrengungen, die Myalgien, Krämpfe, Muskelnekrosen und Myoglobinurie auslösen können. Ein ungewöhnliches pathologisches Merkmal eines PGAM-Mangels ist die Assoziation mit röhrenförmigen Aggregaten. Die Symptome sind eine Unverträglichkeit gegenüber körperlicher Anstrengung oder Aktivität, Krämpfen und Muskelschmerzen. Dauerhafte Schwäche ist selten. Die Krankheit ist nicht fortschreitend und hat eine hervorragende Prognose.
Menschliche Proteine, die diese Domäne enthalten
BPGM ; PFKFB1 ; PFKFB2 ; PFKFB3 ; PFKFB4 ; PGAM1 ; PGAM2 ; PGAM4 ; PGAM5 ; STS1 ; UBASH3A ;
Verweise
Externe Links
- Phosphoglycerat + Mutase an der US National Library of Medicine Medical Subject Headings (MeSH)
- PDBe-KB bietet einen Überblick über alle Strukturinformationen, die im PDB für humane Phosphoglyceratmutase 1 verfügbar sind