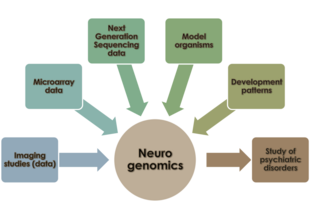
Neurogenomik - Neurogenomics
Neurogenomik ist die Untersuchung, wie das Genom eines Organismus die Entwicklung und Funktion seines Nervensystems beeinflusst. Dieses Feld soll funktionelle Genomik und Neurobiologie vereinen , um das Nervensystem als Ganzes aus einer genomischen Perspektive zu verstehen.
Das Nervensystem von Wirbeltieren besteht aus zwei Haupttypen von Zellen – Neurogliazellen und Neuronen . Beim Menschen gibt es Hunderte von verschiedenen Arten von Neuronen mit unterschiedlichen Funktionen – einige von ihnen verarbeiten äußere Reize; andere erzeugen eine Reaktion auf Reize; andere organisieren sich in zentralisierten Strukturen ( Gehirn , Spinalganglien ), die für die Wahrnehmung, Wahrnehmung und Regulierung motorischer Funktionen verantwortlich sind. Neuronen an diesen zentralisierten Orten neigen dazu, sich in riesigen Netzwerken zu organisieren und intensiv miteinander zu kommunizieren. Vor der Verfügbarkeit von Expressionsarrays und DNA-Sequenzierungsmethoden versuchten die Forscher, das zelluläre Verhalten von Neuronen (einschließlich Synapsenbildung und neuronaler Entwicklung und Regionalisierung im menschlichen Nervensystem) im Hinblick auf die zugrunde liegende Molekularbiologie und Biochemie zu verstehen, ohne jegliches Verständnis von den Einfluss des Genoms eines Neurons auf seine Entwicklung und sein Verhalten. Da sich unser Verständnis des Genoms erweitert hat, hat die Rolle von Netzwerken von Geninteraktionen bei der Aufrechterhaltung der neuronalen Funktion und des neuronalen Verhaltens das Interesse der neurowissenschaftlichen Forschungsgemeinschaft geweckt . Die Neurogenomik ermöglicht es Wissenschaftlern, das Nervensystem von Organismen im Kontext dieser zugrunde liegenden regulatorischen und transkriptionellen Netzwerke zu untersuchen. Dieser Ansatz unterscheidet sich von der Neurogenetik , die die Rolle einzelner Gene ohne Netzwerkinteraktionskontext bei der Untersuchung des Nervensystems betont.
Nähert sich
Aufkommen der Hochdurchsatzbiologie
Im Jahr 1999 berichteten Cirelli & Tononi erstmals über die Assoziation eines genomweiten Genexpressionsprofils im Gehirn (unter Verwendung von Microarrays ) mit einem Verhaltensphänotyp bei Mäusen. Seitdem wurden die globalen Genexpressionsdaten des Gehirns, die von Microarrays abgeleitet wurden, mit verschiedenen verhaltensbezogenen quantitativen Trait-Loci (QTLs) abgeglichen und in mehreren Veröffentlichungen veröffentlicht. Mikroarray-basierte Ansätze haben jedoch ihre eigenen Probleme, die die Analyse durcheinanderbringen – die Sondensättigung kann zu einer sehr kleinen messbaren Varianz der Genexpression zwischen genetisch einzigartigen Individuen führen, und das Vorhandensein von Einzelnukleotidpolymorphismen (SNPs) kann zu Hybridisierungsartefakten führen. Darüber hinaus können Mikroarrays aufgrund ihrer Sonden-basierten Natur viele Arten von Transkripten ( ncRNAs , miRNAs und mRNA- Isoformen ) verpassen . Sonden können auch speziesspezifische Bindungsaffinitäten aufweisen, die eine vergleichende Analyse durcheinanderbringen können.
Insbesondere fällt der Zusammenhang zwischen Verhaltensmustern und einzelnen Gen-Loci mit hoher Penetranz in den Zuständigkeitsbereich der neurogenetischen Forschung, wobei der Fokus darauf liegt, eine einfache kausale Beziehung zwischen einem einzelnen Gen mit hoher Penetranz und einer beobachteten Funktion/einem beobachteten Verhalten zu identifizieren. Es hat sich jedoch gezeigt, dass mehrere neurologische Erkrankungen dazu neigen, polygen zu sein , indem sie von mehreren verschiedenen Genen und regulatorischen Regionen beeinflusst werden, anstatt von einem Gen allein. Es hat daher eine Verschiebung von Einzelgen-Ansätzen zu Netzwerkansätzen zur Untersuchung neurologischer Entwicklung und Erkrankungen stattgefunden, eine Verschiebung, die durch das Aufkommen von Sequenzierungsmethoden der nächsten Generation stark vorangetrieben wurde .
Sequenzierungsansätze der nächsten Generation
Zwillingsstudien haben gezeigt, dass Schizophrenie , bipolare Störung , Autismus-Spektrum-Störung ( ASS ) und Aufmerksamkeits-Defizit-Hyperaktivitäts-Störung (ADHS) hoch vererbbare, genetisch komplexe psychiatrische Störungen sind . Kopplungsstudien sind jedoch weitgehend gescheitert, ursächliche Varianten für solche psychiatrischen Erkrankungen zu identifizieren, vor allem wegen ihrer komplexen genetischen Architektur. Bei betroffenen Personen und Familien können mehrere Varianten mit niedrigem Penetranzrisiko aggregiert werden, und Sätze von ursächlichen Varianten können von Familie zu Familie variieren. Studien in dieser Richtung haben eine polygene Basis für mehrere psychiatrische Erkrankungen festgestellt . Es wurde beispielsweise festgestellt, dass mehrere unabhängig voneinander auftretende de novo-Mutationen bei Patienten mit Alzheimer-Krankheit einen gemeinsamen Satz von funktionellen Signalwegen stören, die an der neuronalen Signalübertragung beteiligt sind. Das Bestreben, die ursächliche Biologie psychiatrischer Erkrankungen zu verstehen, wird daher durch die Fähigkeit, ganze Genome betroffener und nicht betroffener Individuen unvoreingenommen zu analysieren, stark unterstützt.
Mit der Verfügbarkeit von massiv parallelen Sequenzierungsmethoden der nächsten Generation konnten die Wissenschaftler über den sondenbasierten Einfang exprimierter Gene hinausblicken. RNA-seq identifiziert beispielsweise 25-60% mehr exprimierte Gene als Mikroarrays. Auf dem aufstrebenden Gebiet der Neurogenomik hofft man, dass wir durch das Verständnis der genomischen Profile verschiedener Teile des Gehirns unser Verständnis dafür verbessern können, wie die Interaktionen zwischen Genen und Signalwegen die Zellfunktion und -entwicklung beeinflussen. Es wird erwartet, dass dieser Ansatz in der Lage sein wird, die sekundären Gennetzwerke zu identifizieren, die bei neurologischen Erkrankungen gestört sind, und anschließend die Strategie der Medikamentenentwicklung für Hirnerkrankungen unterstützen. Die 2013 ins Leben gerufene BRAIN-Initiative zum Beispiel zielt darauf ab, „ die Entwicklung zukünftiger Behandlungsmethoden für Hirnerkrankungen, einschließlich Alzheimer, Epilepsie und Schädel-Hirn-Trauma , zu informieren “.
Seltene Variantenassoziationsstudien (RVAS) haben die Rolle von De-novo- Mutationen bei mehreren angeborenen und frühkindlichen Erkrankungen wie Autismus hervorgehoben . Einige dieser Protein-zerstörenden Mutationen konnten nur mit Hilfe der gesamten Genomsequenzierung identifiziert und mit RNA-Seq validiert werden . Darüber hinaus sind diese Mutationen nicht in einzelnen Genen statistisch angereichert, sondern zeigen vielmehr Muster statistischer Anreicherung in Gengruppen, die mit Netzwerken verbunden sind, die die neurologische Entwicklung und Aufrechterhaltung regulieren. Eine solche Entdeckung wäre mit bisherigen genzentrischen Ansätzen ( Neurogenetik , Verhaltensneurowissenschaften ) unmöglich gewesen . Die Neurogenomik ermöglicht einen systembasierten Hochdurchsatz-Ansatz zum Verständnis der polygenen Basis neuropsychiatrischer Erkrankungen.
Bildgebende Studien und optisches Mapping
Als in den 1980er Jahren Autismus als eigenständige biologische Störung identifiziert wurde, fanden Forscher heraus, dass autistische Personen in ihren frühen Entwicklungsjahren eine Abnormalität des Gehirnwachstums im Kleinhirn aufwiesen . Spätere Forschungen haben gezeigt, dass 90% der autistischen Kinder im Alter von 2 bis 4 Jahren ein größeres Gehirnvolumen als ihre Altersgenossen haben und eine Zunahme des Gehalts an weißer und grauer Substanz im Großhirn zeigen . Die weiße und graue Substanz im Großhirn wird mit Lernen bzw. Kognition in Verbindung gebracht, und die Bildung von Amyloid-Plaques in der weißen Substanz wird mit der Alzheimer-Krankheit in Verbindung gebracht . Diese Ergebnisse heben den Einfluss der strukturellen Varianz im Gehirn auf psychiatrische Störungen hervor und haben den Einsatz von Bildgebungstechnologien motiviert, um Regionen mit Divergenz zwischen gesunden und kranken Gehirnen zu kartieren. Obwohl es möglicherweise nicht immer möglich ist, biologische Proben aus verschiedenen Bereichen des lebenden menschlichen Gehirns zu gewinnen, bieten Neuroimaging- Techniken ein nichtinvasives Mittel zum Verständnis der biologischen Grundlagen neurologischer Störungen. Es besteht die Hoffnung, dass ein Verständnis der Lokalisierungsmuster verschiedener psychiatrischer Erkrankungen wiederum zu Netzwerkanalysestudien in der Neurogenomik beitragen könnte.
MRT
Strukturelle Magnetresonanztomographie (MRT) kann verwendet werden, um die strukturelle Zusammensetzung des Gehirns zu identifizieren. Insbesondere im Kontext der Neurogenomik hat die MRT in den letzten vier Jahrzehnten eine große Rolle bei der Erforschung der Alzheimer-Krankheit gespielt . Es wurde ursprünglich verwendet, um andere Ursachen von Demenz auszuschließen , aber neuere Studien zeigten das Vorhandensein charakteristischer Veränderungen bei Patienten mit Alzheimer-Krankheit. Aus diesem Grund werden MRT-Scans derzeit als Neuroimaging-Tool verwendet, um die zeitliche und räumliche Pathophysiologie der Alzheimer-Krankheit zu identifizieren, wie z. B. spezifische zerebrale Veränderungen und Amyloid-Bildgebung .
Die einfache und nicht-invasive Natur von MRT-Scans hat Forschungsprojekte motiviert, die die Entwicklung und den Ausbruch psychiatrischer Erkrankungen im Gehirn verfolgen. Die Alzheimer-Krankheit ist zu einem Schlüsselkandidaten in diesem topographischen Ansatz für psychiatrische Erkrankungen geworden. Beispielsweise werden derzeit MRT-Scans verwendet, um die Ruhe- und aufgabenabhängigen Funktionsprofile des Gehirns von Kindern mit autosomal-dominanter Alzheimer-Krankheit zu verfolgen. Diese Studien haben Hinweise auf früh einsetzende Veränderungen des Gehirns bei Risikopersonen für die Alzheimer-Krankheit gefunden. Das Autism Center of Excellence der University of California, San Diego, führt auch MRT-Studien mit Kindern zwischen 12 und 42 Monaten durch, in der Hoffnung, Anomalien der Gehirnentwicklung bei Kindern mit Verhaltenssymptomen von Autismus zu charakterisieren.
Zusätzliche Forschungen haben gezeigt, dass es bei verschiedenen neurologischen Störungen und Erkrankungen spezifische Atrophiemuster im Großhirn (als Folge einer Neurodegeneration ) gibt. Diese krankheitsspezifischen Muster des Fortschreitens der Atrophie können mit MRT-Scans identifiziert werden und liefern einen klinischen Phänotyp-Kontext für die neurogenomische Forschung. Die zeitliche Information über den Krankheitsverlauf, die dieser Ansatz liefert, kann möglicherweise auch die Interpretation von Störungen auf Gennetzwerkebene bei psychiatrischen Erkrankungen beeinflussen.
Optisches Mapping
Ein unerschwingliches Merkmal der Sequenzierungsmethoden der zweiten Generation ist die Obergrenze des genomischen Bereichs, der durch Paarung zugänglich ist . Optical Mapping ist eine aufkommende Methodik, die verwendet wird, um groß angelegte Varianten zu umfassen, die normalerweise nicht mit Paired-End-Reads erkannt werden können. Dieser Ansatz wurde erfolgreich angewendet, um strukturelle Varianten beim Oligodendrogliom , einer Art von Hirntumor, zu erkennen. Jüngste Arbeiten haben auch die Vielseitigkeit optischer Karten bei der Verbesserung bestehender Genomanordnungen hervorgehoben. Chromosomale Umlagerungen , Mikrodeletionen und großflächige Translokationen wurden mit einer beeinträchtigten neurologischen und kognitiven Funktion in Verbindung gebracht , beispielsweise bei hereditärer Neuropathie und Neurofibromatose . Optisches Mapping kann die Erkennung von Varianten signifikant verbessern und Geninteraktionsnetzwerkmodelle für den Krankheitszustand bei neurologischen Erkrankungen informieren.
Studium anderer Hirnerkrankungen
Neben neurologischen Erkrankungen gibt es weitere Erkrankungen, die sich im Gehirn manifestieren und beispielhafte Use-Case-Szenarien für die Anwendung der Bildgebung des Gehirns in der Netzwerkanalyse gebildet haben. Als klassisches Beispiel für bildgebende Genomanalysen verglich eine Forschungsstudie im Jahr 2012 MRT-Scans und Genexpressionsprofile von 104 Gliompatienten, um Behandlungsergebnisse zu unterscheiden und neue zielgerichtete genomische Signalwege bei Glioblastoma Multiforme (GBM) zu identifizieren . Die Forscher fanden zwei verschiedene Gruppen von Patienten mit einer signifikant unterschiedlichen Organisation der weißen Substanz (invasiv vs. nicht-invasiv). Eine anschließende Analyse der Genexpressionsdaten zeigte eine mitochondriale Dysfunktion als den wichtigsten kanonischen Signalweg in einem aggressiven GBM-Phänotyp mit geringer Mortalität.
Die Ausweitung der bildgebenden Verfahren des Gehirns auf andere Krankheiten kann verwendet werden, um andere medizinische Krankheiten bei der Diagnose psychiatrischer Störungen auszuschließen, kann jedoch nicht verwendet werden, um das Vorliegen oder Fehlen einer psychiatrischen Störung zu informieren.
Forschungsentwicklungsmodelle
In Menschen
Die aktuellen Ansätze zur Sammlung von Genexpressionsdaten in menschlichen Gehirnen bestehen darin, entweder Microarrays oder RNA-seq zu verwenden . Derzeit ist es selten, „lebendes“ Gehirngewebe zu entnehmen – nur wenn die Behandlungen eine Gehirnoperation beinhalten, besteht die Möglichkeit, dass während des Eingriffs Gehirngewebe entnommen wird. Dies ist bei Epilepsie der Fall.
Derzeit werden Genexpressionsdaten normalerweise in post mortem Gehirnen gesammelt, und dies ist oft ein Hindernis für die neurogenomische Forschung beim Menschen. Nach dem Tod wird die Zeitspanne zwischen dem Tod und der Erfassung der Daten aus dem post-mortem-Gehirn als Post-Mortem-Intervall (PMI) bezeichnet. Da RNA nach dem Tod abgebaut wird, ist ein frisches Gehirn optimal – aber nicht immer verfügbar. Dies kann wiederum eine Vielzahl von nachgelagerten Analysen beeinflussen. Bei der Arbeit mit Omics-Daten, die von postmortalen Gehirnen gesammelt wurden, sollten die folgenden Faktoren berücksichtigt werden :
- Idealerweise sollte das menschliche Gehirn für eine bestimmte Studie auf PMIs kontrolliert werden.
- Die Todesursache ist auch eine wichtige Variable, die bei der Entnahme von menschlichen Gehirnproben für die Zwecke der Neurogenomik-Forschung zu berücksichtigen ist. Zum Beispiel werden Gehirnproben von Personen mit klinischer Depression oft nach einem Suizid gesammelt. Bestimmte Todesumstände, wie eine Überdosis Drogen oder ein selbstverschuldeter Schuss, verändern den Ausdruck des Gehirns.
- Ein weiteres Problem bei der Untersuchung der Genexpression im Gehirn ist die zelluläre Heterogenität von Hirngewebeproben. Große Gehirnproben können von Fall zu Fall in den Anteilen spezifischer Zellpopulationen variieren. Dies kann die Genexpressionssignaturen beeinflussen und die differentielle Expressionsanalyse signifikant verändern.
- Ein Ansatz, dieses Problem anzugehen, ist die Verwendung von Einzelzell-RNA-seq . Dies würde für einen bestimmten Zelltyp steuern. Diese Lösung ist jedoch nur anwendbar, wenn Studien nicht zelltypspezifisch sind.
Die Differentialdiagnose bleibt auch ein kritischer präanalytischer Störfaktor kohortenweiter Studien zu neurologischen Spektrumserkrankungen. Insbesondere wurde festgestellt, dass dies ein Cromulant-Problem für Studien zur Alzheimer-Krankheit und zu Autismus-Spektrum-Störungen ist. Da sich unser Verständnis der verschiedenen Symptome und der genomischen Grundlagen verschiedener neurogenomischer Erkrankungen verbessert, werden darüber hinaus die diagnostischen Kriterien selbst neu geordnet und überprüft.
Tiermodelle
Die laufende Genomforschung bei neurologischen Erkrankungen neigt dazu, Tiermodelle (und entsprechende Genhomologe ) zu verwenden, um die Netzwerkinteraktionen zu verstehen, die einer bestimmten Erkrankung aufgrund ethischer Probleme bei der Entnahme biologischer Proben aus lebenden menschlichen Gehirnen zugrunde liegen. Auch dies ist nicht ohne Hindernisse.
Die neurogenomische Forschung mit einem Modellorganismus setzt die Verfügbarkeit eines vollständig sequenzierten und annotierten Referenzgenoms voraus. Darüber hinaus müssen die RNA-Profile ( miRNA , ncRNA , mRNA ) des Modellorganismus gut katalogisiert sein, und alle daraus auf den Menschen übertragenen Rückschlüsse müssen eine Grundlage in Funktions-/ Sequenzhomologie haben .
Zebrafisch
Die Entwicklung von Zebrafischen beruht auf Gennetzwerken, die bei allen Wirbeltieren hoch konserviert sind . Darüber hinaus bieten Zebrafische mit einem extrem gut annotierten Satz von 12.000 Genen und 1.000 frühen Entwicklungsmutanten, die tatsächlich in den optisch klaren Zebrafischembryonen und -larven sichtbar sind, ein ausgeklügeltes System für die Mutagenese und Echtzeit-Bildgebung sich entwickelnder Pathologien. Dieses frühe Entwicklungsmodell wurde verwendet, um das Nervensystem mit zellulärer Auflösung zu untersuchen. Das Zebrafisch-Modellsystem wurde bereits verwendet, um Neuroregeneration und schwere polygene menschliche Krankheiten wie Krebs und Herzkrankheiten zu untersuchen. Mehrere Zebrafischmutanten mit Verhaltensvariationen als Reaktion auf die Kokain- und Alkoholdosierung wurden isoliert und können auch eine Grundlage für die Untersuchung der Pathogenese von Verhaltensstörungen bilden.
Nagetier
Nagetiermodelle haben sich bei der Untersuchung menschlicher Erkrankungen bewährt. Diese Modelle wurden ausführlich mit Genhomologen mehrerer monogener Störungen beim Menschen annotiert . Knockout-Studien dieser Homologen haben zu einer Erweiterung unseres Verständnisses der Netzwerkinteraktionen von Genen in menschlichen Geweben geführt. Zum Beispiel wurde das FMR1- Gen aus einer Reihe von Netzwerkstudien mit Autismus in Verbindung gebracht. Die Verwendung eines Knockouts von FMR 1 bei Mäusen schafft das Modell für das Fragile-X-Syndrom , eine der Störungen im Autismus-Spektrum .
Mäuse- Xenotransplantate sind besonders nützlich für die Wirkstoffforschung und waren äußerst wichtig bei der Entdeckung früher antipsychotischer Wirkstoffe. Auch die Entwicklung von Tiermodellen für komplexe psychiatrische Erkrankungen hat sich in den letzten Jahren verbessert. Nagetiermodelle haben Verhaltensänderungen des Phänotyps gezeigt, die einem positiven Schizophrenie- Zustand ähneln , entweder nach genetischer Manipulation oder nach Behandlung mit Medikamenten, die auf Bereiche des Gehirns abzielen, von denen vermutet wird, dass sie Hyperaktivität oder neurologische Entwicklung beeinflussen. Es wurde Interesse an der Identifizierung der durch diese Labormanipulationen vermittelten Netzwerkstörungen geweckt, und die Sammlung genomischer Daten aus Nagetierstudien hat wesentlich zu einem besseren Verständnis der Genomik psychiatrischer Erkrankungen beigetragen.
Das erste Maus-Gehirn- Transkriptom wurde 2008 generiert. Seitdem wurde umfangreiche Arbeit mit dem Aufbau von Modellen für Mäuse mit sozialem Stress geleistet , um die Expressionssignaturen verschiedener psychiatrischer Erkrankungen auf Signalwegebene zu untersuchen. Ein kürzlich erschienener Artikel simulierte die Merkmale der posttraumatischen Belastungsstörung (PTSD) bei Mäusen und erstellte ein Profil des gesamten Transkriptoms dieser Mäuse. Die Autoren fanden eine unterschiedliche Regulation in vielen biologischen Signalwegen, von denen einige mit Angststörungen ( Hyperaktivität , Angstreaktion ), Stimmungsstörungen und Kognitionsstörungen in Verbindung gebracht wurden. Diese Ergebnisse werden durch umfangreiche transkriptomische Analysen von Angststörungen untermauert, und es wird angenommen, dass Veränderungen des Expressionsniveaus in biologischen Signalwegen, die mit Angstlernen und Gedächtnis verbunden sind, zu den Verhaltensmanifestationen dieser Störungen beitragen. Es wird angenommen, dass die funktionelle Anreicherung von Genen, die an der langfristigen synaptischen Potenzierung, Depression und Plastizität beteiligt sind, eine wichtige Rolle beim Erwerb, der Konsolidierung und der Aufrechterhaltung traumatischer Erinnerungen spielt, die Angststörungen zugrunde liegen.
Experimentelle Mausmodelle für psychiatrische Störungen
Ein üblicher Ansatz zur Verwendung eines Mausmodells besteht darin, eine trächtige Maus experimentell zu behandeln, um einen ganzen Wurf zu beeinflussen. Ein zentrales Thema auf diesem Gebiet ist jedoch die Behandlung von Würfen in einer statistischen Analyse. Die meisten Studien berücksichtigen die Gesamtzahl der produzierten Nachkommen, da dies zu einer Erhöhung der statistischen Aussagekraft führen kann. Der richtige Weg ist jedoch, nach der Anzahl der Würfe zu zählen und basierend auf der Wurfgröße zu normalisieren. Es wurde festgestellt, dass mehrere Autismusstudien ihre statistischen Analysen fälschlicherweise auf der Grundlage der Gesamtzahl der Nachkommen statt der Anzahl der Würfe durchgeführt haben.
Mehrere Angststörungen wie die posttraumatische Belastungsstörung (PTSD) beinhalten heterogene Veränderungen in mehreren verschiedenen Gehirnregionen, wie dem Hippocampus , der Amygdala und dem Nucleus accumbens . Es wurde gezeigt, dass die zelluläre Kodierung traumatischer Ereignisse und die durch solche Ereignisse ausgelösten Verhaltensreaktionen hauptsächlich in Veränderungen von Signalmolekülen liegen, die mit der synaptischen Übertragung verbunden sind .
Globale Genexpressionsprofile der verschiedenen Genregionen, die an der Angst- und Angstverarbeitung beteiligt sind, unter Verwendung von Mausmodellen hat zur Identifizierung von zeitlich und räumlich unterschiedlichen Sätzen von unterschiedlich exprimierten Genen geführt. Die Pathway-Analyse dieser Gene hat neben anderen funktionellen und phänotypischen Beobachtungen eine mögliche Rolle bei der Neurogenese und angstbezogenen Verhaltensreaktionen aufgezeigt.
Mäusemodelle für die Hirnforschung haben wesentlich zur Medikamentenentwicklung beigetragen und unser Verständnis der genomischen Grundlagen mehrerer neurologischer Erkrankungen in der letzten Generation verbessert. Chlorpromazin , das erste Antipsychotikum (entdeckt 1951), wurde als praktikable Behandlungsoption identifiziert, nachdem gezeigt wurde, dass es die Reaktion auf aversive Stimuli bei Ratten in einem Verhaltensscreening unterdrückt.
Herausforderungen
Die Modellierung und Bewertung latenter Symptome (Gedanken, verbale Lernprozesse, soziale Interaktionen, kognitives Verhalten) bleibt eine Herausforderung bei der Verwendung von Modellorganismen zur Untersuchung psychiatrischer Erkrankungen mit einer komplexen genetischen Pathologie . Zum Beispiel muss ein gegebener Genotyp + Phänotyp in einem Mausmodell die genomischen Grundlagen eines bei einem Menschen beobachteten Phänotyps imitieren.
Dies ist besonders bei Spektrumstörungen wie Autismus ein entscheidender Punkt . Autismus ist eine Störung, deren Symptome in zwei Kategorien unterteilt werden können: (i) Defizite in sozialen Interaktionen und (ii) sich wiederholende Verhaltensweisen und eingeschränkte Interessen. Da Mäuse eher soziale Wesen unter allen Mitgliedern der Ordnung Rodentia sind, die derzeit als Modellorganismen verwendet werden, werden Mäuse im Allgemeinen verwendet, um menschliche psychiatrische Störungen so genau wie möglich zu modellieren. Insbesondere für Autismus gibt es derzeit die folgenden Workarounds, um menschliche Verhaltenssymptome nachzuahmen:
- Für die erste diagnostische Kategorie von beeinträchtigtem Sozialverhalten werden Mäuse einem Sozialtest unterzogen, der typische autistische soziale Defizite darstellen soll. Zum normalen Sozialverhalten von Mäusen gehören Schnüffeln, Folgen, Körperkontakt und Allogrooming . Auch die Sprachkommunikation kann genutzt werden.
- Es gibt eine Reihe von Möglichkeiten, die zweite diagnostische Kategorie bei Mäusen zu beobachten. Beispiele für sich wiederholendes Verhalten können übermäßiges Kreisen, Selbstpflege und übermäßiges Graben sein. Normalerweise würden diese Verhaltensweisen innerhalb einer langen Zeitdauer (dh Selbstpflege für 10 Minuten) konsequent durchgeführt.
- Während repetitive Verhaltensweisen leicht zu beobachten sind, ist es schwierig, die tatsächlichen eingeschränkten Interessen von Mäusen zu charakterisieren. Ein Aspekt der eingeschränkten Interessen autistischer Personen ist das „Beharren auf Gleichheit“ – das Konzept, dass autistische Personen verlangen, dass ihre Umgebung konsistent bleibt. Wenn sich diese Umgebung ändern sollte, würde die Person Stress und Angst erfahren. Es gibt Berichte über Erfolge bei der Bestätigung eines Mausmodells für Autismus, indem die Umgebung der Maus verändert wurde.
In jedem dieser Experimente haben die „autistischen“ Mäuse einen „normalen“ Sozialpartner und die Wissenschaftler, die die Mäuse beobachten, sind sich der Genotypen der Mäuse nicht bewusst („blind“).
Genexpression im Gehirn
Das Genexpressionsprofil des Zentralnervensystems (ZNS) ist einzigartig. Achtzig Prozent aller menschlichen Gene werden im Gehirn exprimiert; 5.000 dieser Gene werden ausschließlich im ZNS exprimiert. Das menschliche Gehirn weist die höchste Genexpression aller untersuchten Säugetiergehirne auf. Im Vergleich dazu haben Gewebe außerhalb des Gehirns ähnliche Expressionsniveaus im Vergleich zu ihren Gegenstücken bei Säugetieren. Eine Quelle für die erhöhten Expressionsniveaus im menschlichen Gehirn ist die Nicht-Protein-kodierende Region des Genoms. Zahlreiche Studien haben gezeigt, dass das menschliche Gehirn im Vergleich zu anderen Säugetiergehirnen ein höheres Expressionsniveau in regulatorischen Regionen aufweist. Es gibt auch eine bemerkenswerte Bereicherung für weitere alternative Spleißereignisse im menschlichen Gehirn.
Räumliche Unterschiede
Genexpressionsprofile variieren auch innerhalb bestimmter Regionen des Gehirns. Eine Microarray-Studie zeigte, dass sich das Transkriptomprofil der ZNS-Cluster nach Regionen zusammenfügt. Eine andere Studie charakterisierte die Regulation der Genexpression über 10 verschiedene Regionen basierend auf ihren eQTL- Signalen. Die Ursache für die unterschiedlichen Expressionsprofile liegt in Funktion, Neuronenmigration und zellulärer Heterogenität der Region. Sogar die drei Schichten der Großhirnrinde haben unterschiedliche Ausdrucksprofile.
Eine 2014 an der Harvard Medical School abgeschlossene Studie konnte Entwicklungslinien identifizieren, die auf einzelne neuronale Mutationen zurückzuführen sind. Die Forscher sequenzierten 36 Neuronen aus der Großhirnrinde von drei normalen Individuen und stellten fest, dass stark exprimierte Gene und neural assoziierte Gene signifikant für Einzelneuronen- SNVs angereichert waren . Diese SNVs wiederum korrelierten mit Chromatinmarkern der Transkription aus dem fötalen Gehirn.
Entwicklungsmuster beim Menschen
Die Genexpression des Gehirns verändert sich in den verschiedenen Lebensphasen. Die signifikantesten Expressionsniveaus werden während der frühen Entwicklung gefunden, wobei die Genexpressionsrate während der fötalen Entwicklung am höchsten ist. Dies resultiert aus dem schnellen Wachstum von Neuronen im Embryo. Neuronen in diesem Stadium durchlaufen neuronale Differenzierung , Zellproliferation , Migrationsereignisse und dendritische und synaptische Entwicklung . Genexpressionsmuster verschieben sich während der Embryonalentwicklung näher hin zu spezialisierten Funktionsprofilen, jedoch sind bestimmte Entwicklungsschritte bei der Geburt noch im Gange. Folglich erscheinen die Genexpressionsprofile der beiden Gehirnhemisphären bei der Geburt asymmetrisch. Bei der Geburt erscheinen Genexpressionsprofile zwischen den Gehirnhemisphären asymmetrisch . Mit fortschreitender Entwicklung werden die Genexpressionsprofile zwischen den Hemisphären ähnlich. Bei einem gesunden Erwachsenen bleiben die Expressionsprofile von Ende der zwanziger bis in die späten vierziger Jahre relativ konstant. Ab den fünfziger Jahren nimmt die Expression von Genen, die für die normale Funktion wichtig sind, deutlich ab. Trotzdem nimmt die Vielfalt der im Gehirn exprimierten Gene zu. Diese altersbedingte Veränderung der Expression kann mit dem GC-Gehalt korreliert sein . In späteren Lebensstadien kommt es zu einer verstärkten Induktion von Pivotalgenen mit niedrigem GC-Gehalt sowie zu einer verstärkten Repression von Pivotalgenen mit hohem GC-Gehalt. Eine weitere Ursache für die Verschiebung der Genvielfalt ist die Anhäufung von Mutationen und DNA-Schäden. Genexpressionsstudien zeigen, dass Gene, die diese altersbedingten Mutationen verursachen, zwischen Individuen in der alternden Bevölkerung konsistent sind. Gene, die in der Entwicklung stark exprimiert werden, nehmen in den späten Stadien des Lebens signifikant ab, während Gene, die in der Entwicklung stark reprimiert werden, in den späten Stadien signifikant zunehmen.
Evolution des Säugetiergehirns
Die Evolution des Homo sapiens seit der Abweichung vom gemeinsamen Vorfahren der Primaten hat eine deutliche Vergrößerung der Größe und Komplexität des Gehirns, insbesondere der Großhirnrinde, gezeigt . Im Vergleich zu Primaten hat die menschliche Großhirnrinde eine vergrößerte Oberfläche, unterscheidet sich jedoch nur geringfügig in der Dicke. Viele groß angelegte Studien zum Verständnis der Unterschiede des menschlichen Gehirns von anderen Spezies haben gezeigt, dass die Erweiterung von Genfamilien und Veränderungen beim alternativen Spleißen für die damit einhergehende Zunahme der kognitiven Fähigkeiten und des kooperativen Verhaltens beim Menschen verantwortlich sind. Wir müssen jedoch noch die genauen phänotypischen Konsequenzen all dieser Veränderungen bestimmen. Eine Schwierigkeit besteht darin, dass nur Primaten Unterteilungen in ihrer Großhirnrinde entwickelt haben, was es schwierig macht, menschliche spezifische neurologische Probleme bei Nagetieren nachzuahmen .
Sequenzdaten werden verwendet, um die evolutionären genetischen Veränderungen zu verstehen, die zur Entwicklung des menschlichen ZNS führten. Wir können dann verstehen, wie sich die neurologischen Phänotypen zwischen den Arten unterscheiden. Vergleichende Genomik beinhaltet den Vergleich von Sequenzdaten über eine Phylogenie hinweg , um die genotypischen Veränderungen, die innerhalb bestimmter Abstammungslinien auftreten, zu lokalisieren und zu verstehen, wie diese Veränderungen entstanden sein könnten. Die Zunahme qualitativ hochwertiger Referenzsequenzen von Säugetieren verbessert im Allgemeinen die vergleichende Analyse , da sie die statistische Aussagekraft erhöht . Die Zunahme der Artenzahl in einer Phylogenie birgt jedoch die Gefahr, dass unnötiges Rauschen hinzugefügt wird, da die Ausrichtungen der orthologen Sequenzen normalerweise an Qualität verlieren . Darüber hinaus weisen verschiedene Artenklassen signifikante Unterschiede in ihren Phänotypen auf.
Trotzdem hat uns die vergleichende Genomik ermöglicht, die genetischen Veränderungen, die in einer Phylogenie gefunden werden, mit bestimmten Signalwegen zu verbinden. Um dies zu ermitteln, werden Linien auf die im Laufe der Zeit anfallenden funktionalen Veränderungen getestet. Dies wird oft als Verhältnis von nicht synonymen Substitutionen zu synonymen Substitutionen oder als dN/dS-Verhältnis (manchmal weiter abgekürzt als ω) gemessen . Wenn das dN/dS-Verhältnis größer als 1 ist, zeigt dies eine positive Auswahl an . Ein dN/dS-Verhältnis von 1 weist darauf hin, dass keine selektiven Drücke vorhanden sind. Ein dN/dS-Verhältnis von weniger als 1 weist auf eine negative Selektion hin . Beispielsweise weisen die konservierten Regionen des Genoms im Allgemeinen ein dN/dS-Verhältnis von weniger als 1 auf, da jede Änderung dieser Positionen wahrscheinlich schädlich sein wird. Von den im menschlichen Gehirn exprimierten Genen wird geschätzt, dass 342 von ihnen ein dN/dS-Verhältnis von mehr als 1 in der menschlichen Abstammungslinie im Vergleich zu anderen Primatenlinien aufweisen. Dies zeigt eine positive Selektion auf die menschliche Abstammungslinie für Gehirn-Phänotypen an. Die Bedeutung der positiven Selektion zu verstehen ist im Allgemeinen der nächste Schritt. Zum Beispiel ASPM , CDK5RAP2 und NIN sind Gene , die für auf der menschlichen Linie positiv selektiert sind und mit Gehirngröße direkt korreliert. Dieser Befund könnte helfen zu erklären, warum menschliche Gehirne größer sind als andere Säugetiergehirne.
Expressionsunterschiede auf Netzwerkebene zwischen Arten
Es wird angenommen, dass Veränderungen der Genexpression, die die ultimative Reaktion auf genetische Veränderungen sind, ein guter Proxy für das Verständnis phänotypischer Unterschiede innerhalb biologischer Proben sind. Vergleichende Studien haben eine Reihe von Unterschieden in der Transkriptionskontrolle zwischen Primaten und Nagetieren gezeigt . Beispielsweise ist das Gen CNTNAP2 im präfrontalen Kortex spezifisch angereichert . Das Maushomologe von CNTNAP2 wird im Mausgehirn nicht exprimiert. CNTNAP2 wurde in der kognitiven Funktionen der Sprache sowie verwickelt neurologische Entwicklungsstörungen wie Autismus - Spektrum - Störung. Dies legt nahe, dass die Kontrolle der Expression eine bedeutende Rolle bei der Entwicklung der einzigartigen menschlichen kognitiven Funktion spielt. Infolgedessen haben eine Reihe von Studien die gehirnspezifischen Enhancer untersucht. Es wurde festgestellt, dass Transkriptionsfaktoren wie SOX5 in der menschlichen Abstammungslinie positiv selektiert werden. Genexpressionsstudien bei Menschen, Schimpansen und Rhesusaffen haben humanspezifische Co-Expressionsnetzwerke und eine Erhöhung der Genexpression im menschlichen Kortex im Vergleich zu Primaten identifiziert.
Störungen
Neurogenomische Störungen manifestieren sich als neurologische Störungen mit einer komplexen genetischen Architektur und einem nicht-Mendelschen Vererbungsmuster. Einige Beispiele für diese Störungen umfassen bipolare Störungen und Schizophrenie . An der Manifestation der Erkrankung können mehrere Gene beteiligt sein, und Mutationen bei solchen Erkrankungen sind im Allgemeinen selten und neu . Daher wird es äußerst unwahrscheinlich, dieselbe (potenziell ursächliche) Variante bei zwei nicht verwandten Personen zu beobachten, die von derselben neurogenomischen Störung betroffen sind. Laufende Forschungen haben zum Beispiel mehrere de novo exonische Variationen und strukturelle Variationen bei der Autismus-Spektrum-Störung ( ASS) impliziert . Das allelische Spektrum der seltenen und häufigen Varianten bei neurogenomischen Erkrankungen erfordert daher einen Bedarf an großen Kohortenstudien, um Varianten mit geringer Wirkung effektiv auszuschließen und die übergreifenden Signalwege zu identifizieren, die bei den verschiedenen Erkrankungen häufig mutiert sind, und nicht spezifische Gene und spezifische Mutationen mit hoher Penetranz .
Whole Genome Sequencing (WGS) und Whole Exom Sequencing (WES) wurden in Genome Wide Association Studies (GWAS) verwendet, um genetische Varianten im Zusammenhang mit neurogenomischen Störungen zu charakterisieren. Die Auswirkungen dieser Varianten können jedoch aufgrund der bei mehreren dieser Erkrankungen beobachteten nicht-Mendelschen Vererbungsmuster nicht immer überprüft werden. Ein weiteres unerschwingliches Merkmal der Netzwerkanalyse ist der Mangel an groß angelegten Datensätzen für viele psychiatrische (neurogenomische) Erkrankungen. Da mehrere Erkrankungen mit neurogenomischen Grundlagen dazu neigen, polygenetisch zu sein, können mehrere unspezifische, seltene und teilweise penetrante De-novo- Mutationen bei verschiedenen Patienten zu derselben beobachteten Bandbreite von Phänotypen beitragen, wie dies bei der Autismus-Spektrum-Störung und der Schizophrenie der Fall ist. Umfangreiche Forschung in Alkoholabhängigkeit hat auch die Notwendigkeit einer qualitativ hochwertige Genomprofilerstellung von großen Probensätzen hervorgehoben , wenn das Studium polygener , Spektrum - Störungen .
Das 1000-Genome-Projekt war eine erfolgreiche Demonstration dafür, wie eine konzertierte Anstrengung zur Erfassung repräsentativer Genomdaten aus dem breiten Spektrum des Menschen zur Identifizierung umsetzbarer biologischer Erkenntnisse für verschiedene Krankheiten führen kann. Gerade im Bereich der neurogenomischen Erkrankungen fehlt jedoch eine solche groß angelegte Initiative.
Modellierung psychiatrischer Störungen in der Neurogenomikforschung – Fragen
Eine große GWAS- Studie identifizierte 13 neue Risikoorte für Schizophrenie . Die Untersuchung der Auswirkungen dieser Kandidaten würde idealerweise einen Schizophrenie-Phänotyp in Tiermodellen zeigen, der aufgrund seiner Manifestation als latente Persönlichkeit normalerweise schwer zu beobachten ist. Dieser Ansatz ist in der Lage, den molekularen Einfluss des Kandidatengens zu bestimmen . Idealerweise hätten die Kandidatengene einen neurologischen Einfluss, was wiederum darauf hindeutet, dass sie bei der neurologischen Störung eine Rolle spielen. In der oben erwähnten GWAS-Studie zur Schizophrenie stellten Ripke und Kollegen beispielsweise fest, dass diese Kandidatengene alle an der Calciumsignalübertragung beteiligt sind. Alternativ kann man diese Varianten in Modellorganismen im Kontext der betroffenen neurologischen Funktion untersuchen. Es ist wichtig zu beachten, dass die Varianten dieser Erkrankungen mit hoher Penetranz dazu neigen, de novo- Mutationen zu sein.
Eine weitere Komplikation bei der Untersuchung neurogenomischer Störungen ist die heterogene Natur der Störung. Bei vielen dieser Erkrankungen bleiben die von Fall zu Fall beobachteten Mutationen nicht konsistent. Bei Autismus kann eine betroffene Person eine große Anzahl schädlicher Mutationen im Gen X erfahren. Eine andere betroffene Person hat möglicherweise keine signifikanten Mutationen in Gen X, aber eine große Anzahl von Mutationen in Gen Y. Die Alternative besteht darin, zu bestimmen, ob Gen X und Gen Y beeinflussen denselben biochemischen Weg – einen, der eine neurologische Funktion beeinflusst. Eine bioinformatische Netzwerkanalyse ist ein Ansatz für dieses Problem. Netzwerkanalysemethoden bieten einen verallgemeinerten Systemüberblick über einen molekularen Stoffwechselweg.
Eine letzte zu berücksichtigende Komplikation ist die Komorbidität neurogenomischer Gene. Mehrere Störungen, insbesondere an den schwereren Enden des Spektrums, neigen dazu, miteinander komorbid zu sein. Schwerere Fälle von ASS sind beispielsweise in der Regel mit geistiger Behinderung (ID) verbunden. Dies wirft die Frage auf, ob es echte, einzigartige ASD-Gene und einzigartige ID-Gene gibt oder ob es nur Gene gibt, die nur mit neurologischen Funktionen in Verbindung gebracht werden und zu einem abnormalen Phänotyp mutiert werden können. Ein Störfaktor kann die tatsächliche Diagnosekategorie und die Methoden des Spektrums der Störungen sein, da die Symptome zwischen schweren Störungen ähnlich sein können. Eine Studie untersuchte die komorbiden Symptome zwischen Gruppen von ID und ASD und fand keinen signifikanten Unterschied zwischen den Symptomen von ID-Kindern, ASD-Kindern mit ID und ASD-Kindern ohne ID. Zukünftige Forschung kann dazu beitragen, eine stringentere genetische Grundlage für die Diagnose dieser Erkrankungen zu schaffen.
Netzwerkanalyse
Das Hauptziel der Netzwerkanalyse in der Neurogenomik besteht darin, statistisch signifikante nicht-zufällige Assoziationen zwischen Genen zu identifizieren, die Risikovarianten enthalten. Obwohl es bereits mehrere Algorithmusimplementierungen dieses Ansatzes gibt, bleiben die allgemeinen Schritte für die Netzwerkanalyse gleich.
- Der analytische Prozess beginnt mit der Identifizierung eines biologischen Netzwerks auf der Grundlage einer experimentellen Validierung. Dies kann ein Gen-Co-Expressionsnetzwerk oder ein Protein-Protein-Interaktions- (PPI)-Netzwerk sein. Die Knoten des Netzwerks werden geclustert.
- Anschließend wird eine spezifische Liste von Genen mit bekannten Assoziationen zu einem bestimmten interessierenden Phänotyp erstellt. Diese Liste könnte durch experimentelle Daten bestimmt werden, die unabhängig von genetischen Studien bei psychiatrischen Erkrankungen sind. Dies wird als 'Trefferliste' bezeichnet.
- Gene, die zur Trefferliste gehören, sowie das im ersten Schritt ausgewählte biologische Netzwerk werden als solche gekennzeichnet.
- Darauf folgt ein Schuld-durch-Assoziations-Schritt (GBA). Dies bedeutet, dass Cluster innerhalb des biologischen Netzwerks, die eine signifikante Menge an Genen aus der Trefferliste aufweisen, mithilfe von Tools zur funktionellen Anreicherung und Datenbankabfragen nach den Pfaden, an denen diese hoch bewerteten Cluster-Gene beteiligt sind, weiter untersucht werden
- Auf diese Weise werden die biologischen Assoziationen der hochbewerteten, experimentell implizierten Clustermitglieder untersucht, wobei der Suchbereich über die anfängliche Trefferliste hinaus erweitert wird, um Genmitglieder zusätzlicher Signalwege einzubeziehen, die möglicherweise eine signifikante Assoziation mit dem betrachteten anfänglichen biologischen Netzwerk haben. Dies führt zu einer Reihe von Kandidatengenen.
Das zugrunde liegende Prinzip dieses Ansatzes ist, dass die Gene, die sich zusammenschließen, auch gemeinsam denselben molekularen Weg beeinflussen. Auch hier wären sie idealerweise Teil einer neurologischen Funktion. Die Kandidatengene können dann verwendet werden, um Varianten für die Nasslaborvalidierung zu priorisieren.
Neuropharmakologie
Historisch gesehen beruhten die Therapien aufgrund der Verhaltensstimulation, die sich als Symptom bei mehreren neurogenomischen Störungen manifestierte, hauptsächlich auf Antipsychotika oder Antidepressiva. Diese Medikamentenklassen würden häufige Symptome der Störungen unterdrücken, jedoch mit fragwürdiger Wirksamkeit. Das größte Hindernis für die neuropharmakogenomische Forschung waren die Kohortengrößen. Angesichts der neu verfügbaren Sequenzierungsdaten großer Kohorten gab es in letzter Zeit einen Vorstoß, die therapeutischen Optionen zu erweitern. Die Heterogenität neurologischer Erkrankungen ist die Hauptmotivation für personalisierte medizinische Ansätze ihrer Therapien. Es ist selten, einzelne ursächliche Gene mit hoher Penetranz bei neurologischen Erkrankungen zu finden. Die genomischen Profile variieren verständlicherweise von Fall zu Fall, und logischerweise müssten die Therapien zwischen den Fällen variieren. Erschwerend kommt hinzu, dass viele dieser Störungen Spektrumstörungen sind. Ihre genetische Ätiologie wird innerhalb dieses Spektrums variieren. Zum Beispiel ist schwere ASS mit De-novo-Mutationen mit hoher Penetranz verbunden. Leichtere Formen von ASS sind normalerweise mit einer Mischung aus häufigen Varianten verbunden.
Das Schlüsselthema ist dann die Übersetzung dieser neu identifizierten genetischen Varianten (aus Studien mit Kopienzahlvarianten , Kandidatengensequenzierung und Hochdurchsatz-Sequenzierungstechnologien) in eine Intervention für Patienten mit neurogenomischen Störungen. Ein Aspekt wird sein, ob die neurologische Störung medizinisch behandelbar ist (dh gibt es einen einfachen Stoffwechselweg, auf den eine Therapie abzielen kann). Zum Beispiel wurden spezifische Fälle von ASD mit Mikrodeletionen des TMLHE- Gens in Verbindung gebracht. Dieses Gen kodiert für das Enzym der Carnitin-Biosynthese . Nahrungsergänzungsmittel zur Erhöhung des Carnitinspiegels schienen bestimmte ASS-Symptome zu lindern, aber die Studie wurde durch viele Einflussfaktoren verfälscht. Wie bereits erwähnt, wird die Verwendung eines Gennetzwerk-Ansatzes dazu beitragen, relevante Pfade von Interesse zu identifizieren. Viele neuropharmakogenomische Ansätze haben sich auf die nachgelagerten Produkte dieser Stoffwechselwege konzentriert.
Blut-Hirn-Schranke
Studien an Tiermodellen für verschiedene Hirnerkrankungen haben gezeigt, dass die Blut-Hirn-Schranke (BBB) auf vielen Ebenen Veränderungen unterliegt; zum Beispiel kann die Zusammensetzung der Oberflächenglykoproteine die Arten von HIV-1-Stämmen beeinflussen, die von der BBB transportiert werden. Es wurde festgestellt, dass die BBB eine Schlüsselrolle bei der Entstehung der Alzheimer- Krankheit spielt. Es ist jedoch äußerst schwierig, dies am Menschen zu untersuchen, da der Zugang zum Gehirn und die Entnahme biologischer Proben für die Sequenzierung oder morphologische Analyse offensichtlich eingeschränkt sind. Mäusemodelle der BHS und Modelle von Krankheitszuständen haben gute Dienste geleistet, um die BHS als regulatorische Schnittstelle zwischen Krankheit und guter Gesundheit im Gehirn zu konzeptualisieren.
Personalisierte Neurobiologie
Die Heterogenität neurologischer Erkrankungen ist die Hauptmotivation für personalisierte medizinische Ansätze für ihre Therapien. Genomische Proben einzelner Patienten könnten verwendet werden, um prädiktive Faktoren zu identifizieren oder die spezifische Prognose einer neurogenomischen Erkrankung besser zu verstehen und diese Informationen als Leitfaden für Behandlungsoptionen zu verwenden. Obwohl dieser Ansatz einen klaren klinischen Nutzen hat, ist die Adaption dieses Ansatzes immer noch nicht vorhanden.
Es gibt verschiedene Probleme, die die Anwendung personalisierter Genomik zur Beurteilung, Diagnose und Behandlung psychiatrischer Störungen verbieten.
- Erstens ist die ursächliche Netzwerkbiologie mehrerer Spektrumserkrankungen mit neurogenomischen Grundlagen noch nicht vollständig verstanden, trotz umfangreicher Studien, die mit Erkrankungen wie Autismus-Spektrum und Schizophrenie durchgeführt wurden . Somit ist die analytische Validität von bestehenden Hypothesen zur Ätiologie neurogenomischer Erkrankungen noch nicht vollständig geklärt und wird diskutiert und kontrovers diskutiert.
- Die klinische Validität genetischer Varianten, die nachweislich stark mit bestimmten neurogenomischen Erkrankungen korreliert sind, gibt häufig Anlass zu großer Besorgnis. Die Interpretation dieser Testergebnisse und die anschließende Entscheidungsfindung sind angesichts der polygenen Natur vieler dieser Erkrankungen ein kompliziertes Unterfangen. Erschwerend kommt hinzu, dass sich gezeigt hat, dass präventive Interventionen bei schwerwiegenden psychiatrischen Störungen das Risiko für die Störung nicht immer verringern. Eine solche Intervention ist möglicherweise nicht einmal für gefährdete Nachkommen betroffener Erwachsener verfügbar, wodurch die „medizinische Verwertbarkeit“ der Daten eingeschränkt wird.
- Es wurden auch ethische Bedenken hinsichtlich des Schutzes persönlicher genomischer Informationen und des Umgangs mit der Belastung durch Zufallsbefunde und der Risikobewertung der Familie geäußert.
- Blutsverwandtschaft und Inzucht können zu einer selektiven Anreicherung seltener genetischer Mutationen mit ansonsten geringer Penetranz führen, die auf verschiedene Symptome neurogenomischer Erkrankungen zurückzuführen sind. Daher erfordert die Interpretation familienspezifischer genetischer Mutationen und/oder Störungen auf Netzwerkebene beim Auftreten einer seltenen psychiatrischen Störung eine sorgfältige Abwägung der Motivation der in die Studie eingeschlossenen Teilnehmer.
- Allerdings können diese Probleme durch eine wirksame Aufklärung und Beratung angegangen werden, und die Sammlung genomischer Daten von Patienten mit psychiatrischen Erkrankungen sollte nicht allein auf dieser Grundlage ausgeschlossen werden. Die Daten selbst dienen als dynamische Gesundheitsressource und können unser Verständnis der genomischen Grundlagen mehrerer psychiatrischer Erkrankungen erheblich verbessern.